Construindo uma árvore de máxima verossimilhança com o software IQ-TREE3
Aproveitando a aula da disciplina de Biogeografia, hoje faremos um tutorial para construção de uma árvore de máxima verossimilhança e, para isso, vamos utilizar o programa IQ-TREE3. Apesar de haver alguns software que são normalmente utilizados para inferência baseada em máxima verossimilhança, este é um dos que eu mais gosto pois é um programa rápido e bastante completo. Antes de começar, faça o download do programa no website, de acordo com seu sistema operacional. Neste tutorial, usaremos Windows 11 e, se você usa o mesmo sistema e quer ir direto ao ponto, pode baixá-lo aqui.
Primeiramente, precisamos de um alinhamento. Vamos utilizar a matriz de um fragmento de mtDNA (ou DNA mitocondrial) do gene 16S, do trabalho em que Taucce et al. (2020) descrevem o anuro Pristimantis rupicola. A matriz já está alinhada e pronta para ser utilizada. Agora, descompacte o arquivo que você baixou do site do IQ-TREE2 e coloque a matriz p_rupícola.fasta dentro da pasta com os arquivos descompactados, em uma pasta com o programa propriamente dito, chamada bin.
Depois de tudo pronto, a primeira coisa que vamos fazer é clicar no programa, que está na pasta bin. Clique na versão do programa chamada iqtree3-click.exe. Você verá uma janela assim:
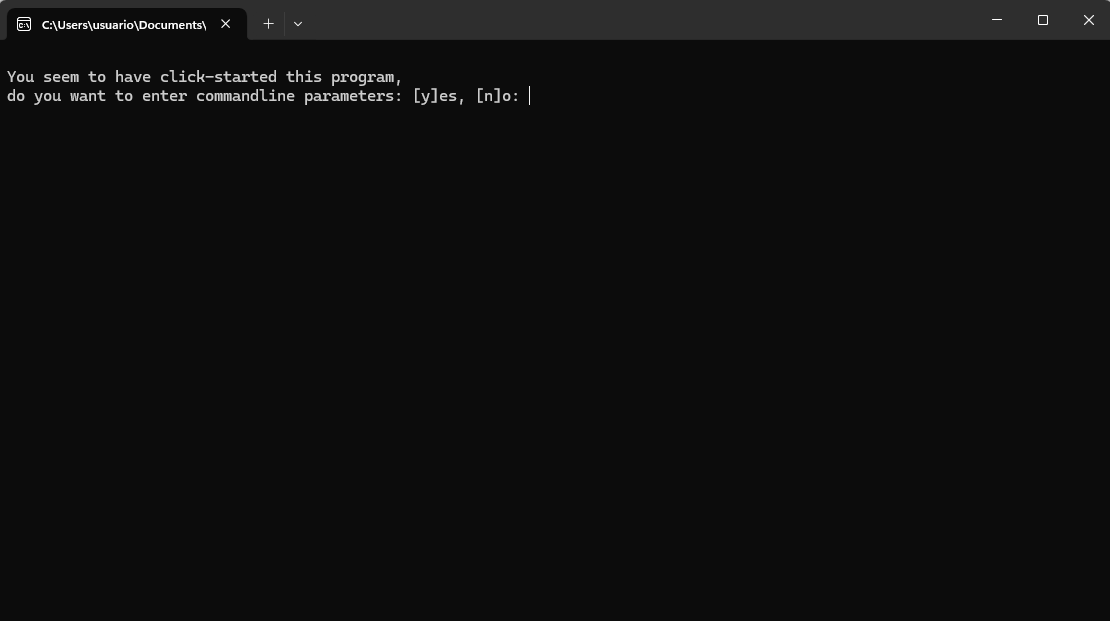
Digite y e aperte enter. Você verá uma mensagem pedindo para que possamos inserir os parâmetros. Para realizar uma análise de máxima verossimilhança, primeiramente precisaremos inferir o melhor modelo de substituição nucleotídica. No entanto, o IQ-TREE pode fazer isso de forma automática, logo antes da busca pela árvore mais verossímil. Isso salva um monte de tempo, então vamos usar! Faremos então a busca pelo modelo de substituição nucleotídica que mais se encaixa nos dados, depois a busca pela árvore mais verossímil seguida de 1000 réplicas de bootstrap, para que possamos saber o nível de suporte dos nossos clados. Para isso, vamos digitar um comando de cada vez. Primeiro, -s, e então, enter. Esse primeiro comando significa que vamos fazer a busca pelo melhor modelo e então pela melhor árvore. Aperte e de extend, enter e então escreva o nome do arquivo, que está na pasta, no nosso caso p_rupicola.fasta, e então enter de novo. Depois digite e, aperte enter e escreva -B, comando para as réplicas de bootstrap ultrarápido, implementado no IQ-TREE, e aperte enter. Depois de apertar e novamente, digite o número de réplicas de bootstrap: 1000 está de bom tamanho. Aperte enter e então sua tela deve estar assim:
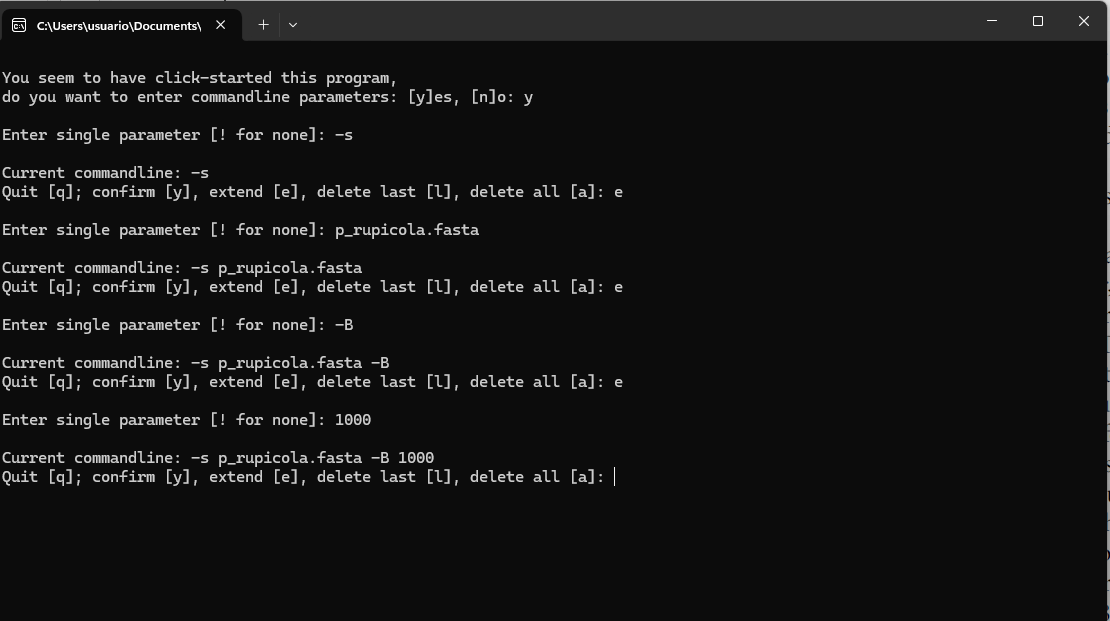
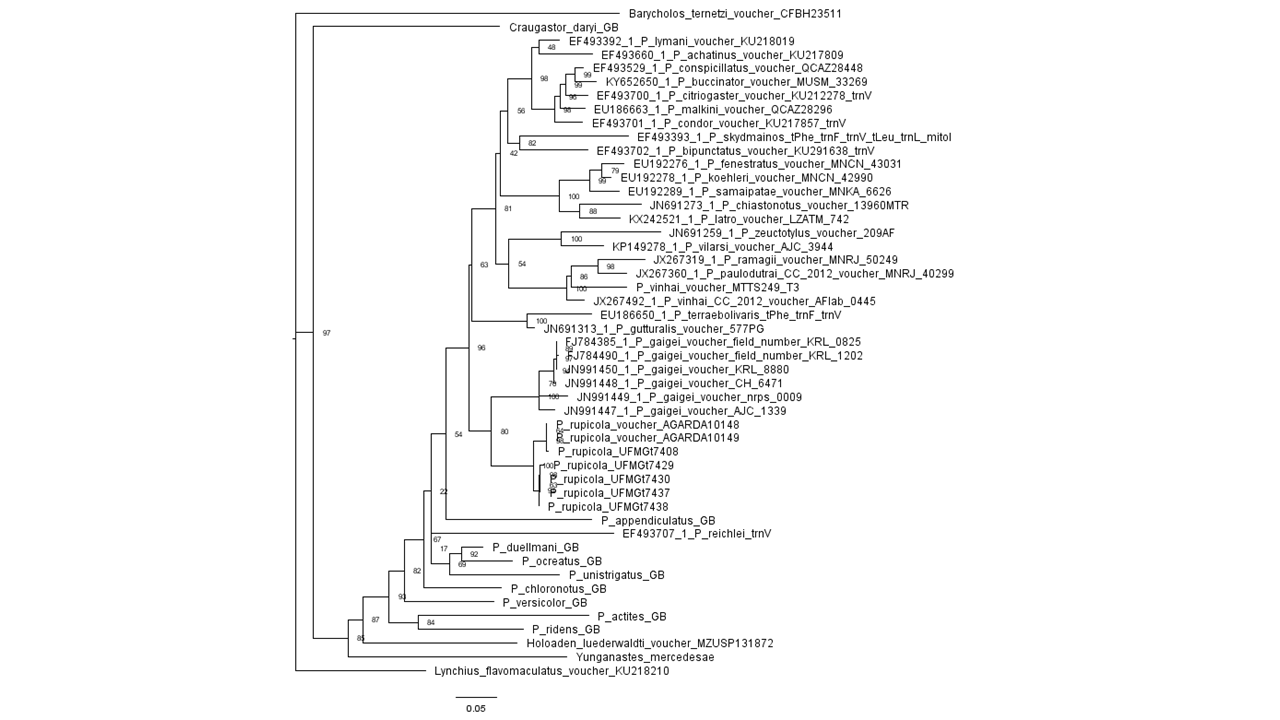
Se tudo deu certo, confirme apertando y e então enter. Sua análise vai começar a rodar! Depois de tudo acabado, é só fechar a janela e dar uma olhada na pasta. Dentre os vários arquivos, está a nossa árvore: p_rupicola.fasta.treefile. Pronto! Agora é só usar um visualizador de árvore como por exemplo o FigTree e comparar os resultados com a minha árvore!
Enjoy Reading This Article?
Here are some more articles you might like to read next: